GraphCare: Enhancing Healthcare Predictions with Open-World Personalized Knowledge Graphs
文章: ArXiv
代码: Github
日期: 2023.05.22
作者: Pengcheng Jiang, Cao Xiao, Adam Cross, Jimeng Sun
机构: UIUC, Relativity, OSF HealthCare
研究背景和动机
常见的健康预测任务有:死亡预测,住院时长预测,再次住院预测,药物推荐等。
为了提升根据电子病历数据做预测的表现,同时把专家知识和数据洞见结合起来,临床知识图谱(KG)就是一个重要的知识来源。KG中包括医疗概念(如,诊断、治疗、药物等)和它们之间的关联。
- 现有方法的局限:主要关注实体间的层次关系(比如ICD9或者ICD10编码之间的层级关系),没有充分挖掘KG中各种实体间的复杂关系以学习更多上下文的知识;大语言模型(LLMs)已经展现出作为知识库的潜力,可以用作额外的临床知识提取器(还没有相关的尝试)。
- 新的想法:提出了个性化医疗KG的概念(personalized medical KG, or patient-specific KG),并利用LLM提取并形成个性化KG,以有效利用丰富的临床知识。
方法
作者设计了GraphCare方法,实现从自动化知识获取到利用知识进行健康预测的这个流程,主要包括三个步骤:
- 概念知识图谱的构建:临床数据中有医疗实体,需要为它们找到关联的实体以构建知识图
- 个性化医疗KG:每个患者的电子病历中出现的医疗实体可以构成自己的KG,确定诊断/治疗等实体对最终预测的影响
- 预测模型设计:每个患者有自己的KG,怎么根据这个图做预测
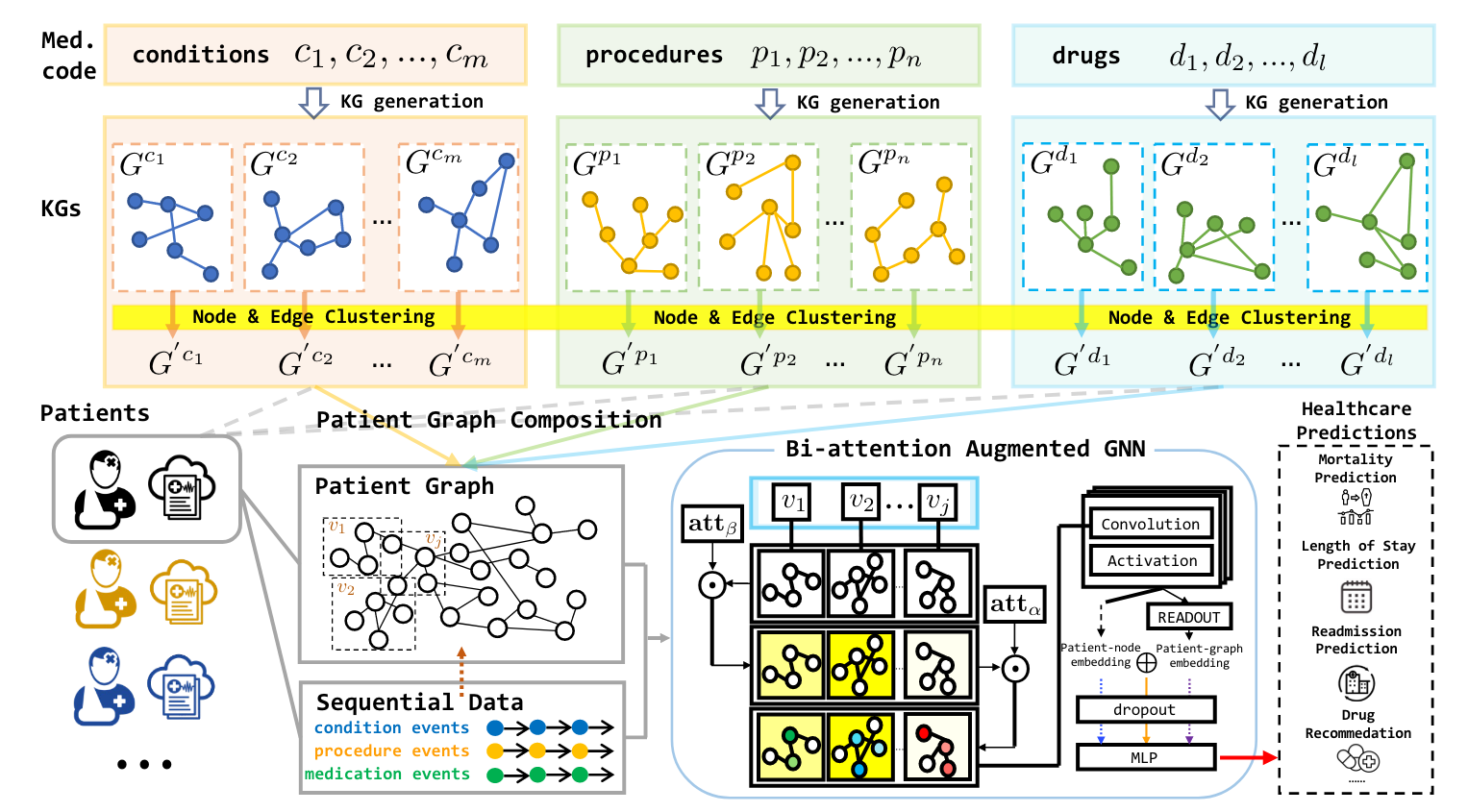
图1. GraphCare 框架图
概念KG生成(Concept-specific KG)
2个来源:大模型(参数知识)和现有的医疗知识图谱(明确知识)
主要有三种类型的实体2:
- condition:诊断
- procedure:治疗的方式和手段
- drug:用药

图2. Medical Code 示例
根据Prompt从大模型抽取
核心:怎么写提示更好(给LLM示例和明确的输出要求,如图 3);作者重复跑了3次以获取更多KG信息。
收集的KG规模为:42,056个实体、9,404种关系、85,387个三元组

图3. LLM根据提示生成KG
从现有知识图谱抽取
使用了英文医疗知识图谱有UMLS1,从中随机采样了每个实体2-hop的三元组。
收集的KG规模为:82,628个实体、80种关系、247,069个三元组

图4. UMLS采样
节点和边聚类
获取的KG中包含大量相似的实体和关系,规模较大,所以需要先做聚类以简化后续模型训练过程的复杂性。聚类算法使用层次聚类(agglomerative clustering algorithm),节点和边的特征为它们名称的 word embedding(从LLM获取)。
通过聚类,可以将一些节点以及一些边组合到一起,因此新 KG 的节点和边就是原始 KG 节点和边的 cluster。它们的 word embedding 是 cluster 内所有成员 word embedding 的平均值。
患者个性化 KG 生成 (Personalized KG)
图构建方法:将一个患者当作一个节点,和他电子病历中出现的所有诊断/治疗/用药的实体相连,再加上这些实体对应的 Concept-specific KGs。
对于患者 i, 有 J 次就诊记录,则可以表示成
Gpat(i)={Gi,1,Gi,2,⋯,Gi,J}={(Vi,1,Ei,1),⋯,(Vi,J,Ei,J)}其中,V 和 E 分别表示图的节点和边。
双注意力增强的图神经网络 (BAT)
经典 GNN 模型
图神经网络的基本思路为:
- 信息传递:将邻居节点的信息传递过来
- 聚合:将自身和邻居的信息做聚合
- 更新:更新自身的表示

图5. 图神经网络的基本思路
数学表达形式为
hk(l+1)=σ(W(l)AGGREGATE(l)(hk′(l)∣k′∈N(k))+b(l))
- hk(l+1): 节点 k 的在第 (l+1) 层更新的节点表示
- AGGREGATE(l):第 l 层邻居节点信息的聚合函数
- W(l) 和 b(l): 待学习参数
- σ:激活函数
局限:患者的个性化 KG Gpat(i) 是时序图数据,带有时序信息;经典 GNN 不能很好地捕捉到复杂变化信息。
双注意力GNN (Bi-attention Augmented GNN, BAT)
作者从2个层次上提出注意力机制:
1. 多次就诊记录的重要程度(subgraph-level) ⟶βi,j (患者 i 的第 j 次就诊的权重)
βi,1,...,βi,N=λ⊤Tanh(wβ⊤Gi+bβ),whereλ=[λ1,...,λN]
- λ∈RN, 是时间衰减向量
- λj=e−γ(J−j),j≤J,即随着j→J, λj值更大(最近的就诊更重要)
- Gi∈RN×M,M是所有患者电子病历中出现的医疗实体数量,N是所有患者中最大的就诊次数
- Gi,j,k=1说明患者 i 的第 j 次就诊包含第 k 个医疗实体,否则为0
2. 一次就诊中某个诊断/治疗/用药的重要程度(node-level) ⟶αi,j,k (患者 i 的第 j 次就诊的第 k 个节点的权重)
αi,j,1,...,αi,j,M=Softmax(Wαgi,j+bα)
- gi,j∈RM 记录患者 i 第 j 次就诊包括的医疗实体
BAT的聚合更新方式:邻居节点 + 边信息
hi,j,k(l+1)=σ⎝⎛W(l)j′∈J,k′∈N(k)∪{k}∑(αi,j′,k(l)βi,j′(l)hi,j′,k′(l)+wR<k,k′>(l)h(i,j,k)↔(i,j′,k′))+b(l)⎠⎞注意力初始化:Wα反映的是每个医疗实体(即node)对于健康预测任务的重要性,所以初始化的时候可以用word embedding计算。比如,对预测死亡的任务,可以计算每个实体的 embedding 和 “death” 这个词的 embedding 之间的 cosine 相似度。
患者的表示:经过 L 层 BAT 后
- 所有就诊记录涉及到的node取平均(整个图取平均):
hiGpat=MEAN(j=1∑Jk=1∑Kjhi,j,k(L))
- 所有就诊记录中和患者直接相连的node取平均(针对患者这个节点的邻居取平均):
hiP=MEAN(j=1∑Jk=1∑Kj1i,j,kΔhi,j,k(L))
zigraph=MLP(hiGpat)zinode=MLP(hiP)zijoint=MLP(hiGpat⊕hiP)训练和预测
任一患者有 t 次就诊记录,则可以表示成{(x1),(x1,x2),...,(x1,x2,...,xt)},每个 tuple 包含当前就诊之前的历史记录
- 死亡预测(二分类),根据之前的就诊记录预测本次的生存情况,即f:(x1,x2,...,xt−1)→y[xt],y[xt]∈{0,1}
- 再次入院预测(二分类),根据之前的就诊记录预测未来15天内是否再次入院,即f:(x1,x2,...,xt−1)→y[τ(xt)−τ(xt−1)],y∈{0,1}
- 住院时长预测(多分类),预测本次就诊的住院时长,即f:(x1,x2,...,xt)→y[xt],y[xt]∈R1×C,分成10类(0-7天[8类],1-2周,超过2周)
- 用药推荐(多标签),预测本次就诊使用的药物,即f:(x1,x2,...,xt)→y[xt],y[xt]∈R1×∣d∣
实验
数据
- 医疗数据:MIMIC-III、MIMIC-IV
- LLM:GPT-4
- 医疗知识图谱:UMLS
- 检索word embedding:GPT-3
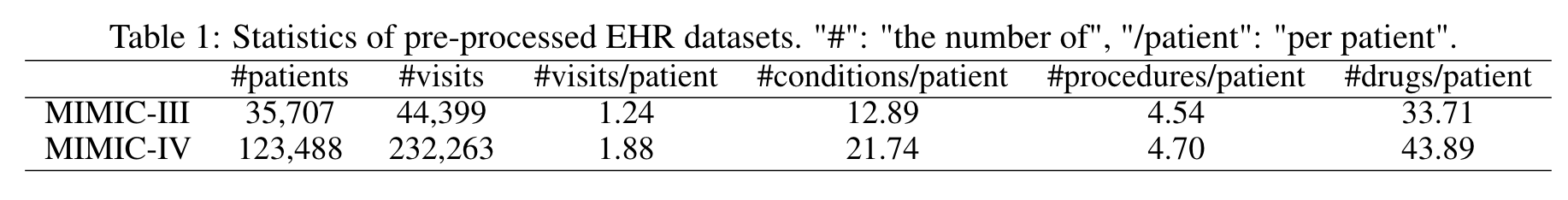
图6. 数据说明
主结果
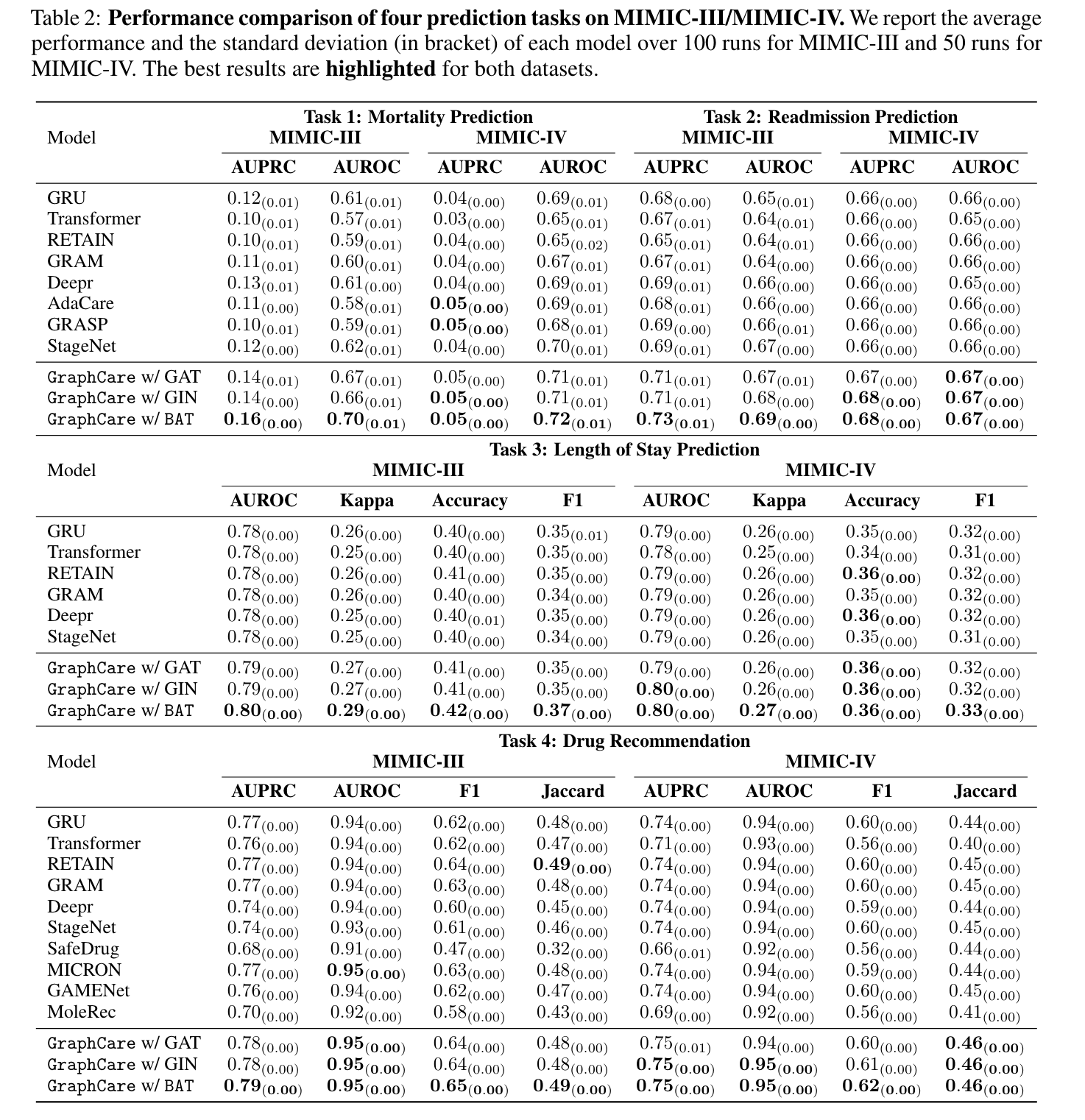
图7. 主要结果
个性化KG的分析
训练数据量的比较
- 训练数据越多,效果越好
- GraphCare在数据量较少的情况下,能得到较好的预测结果
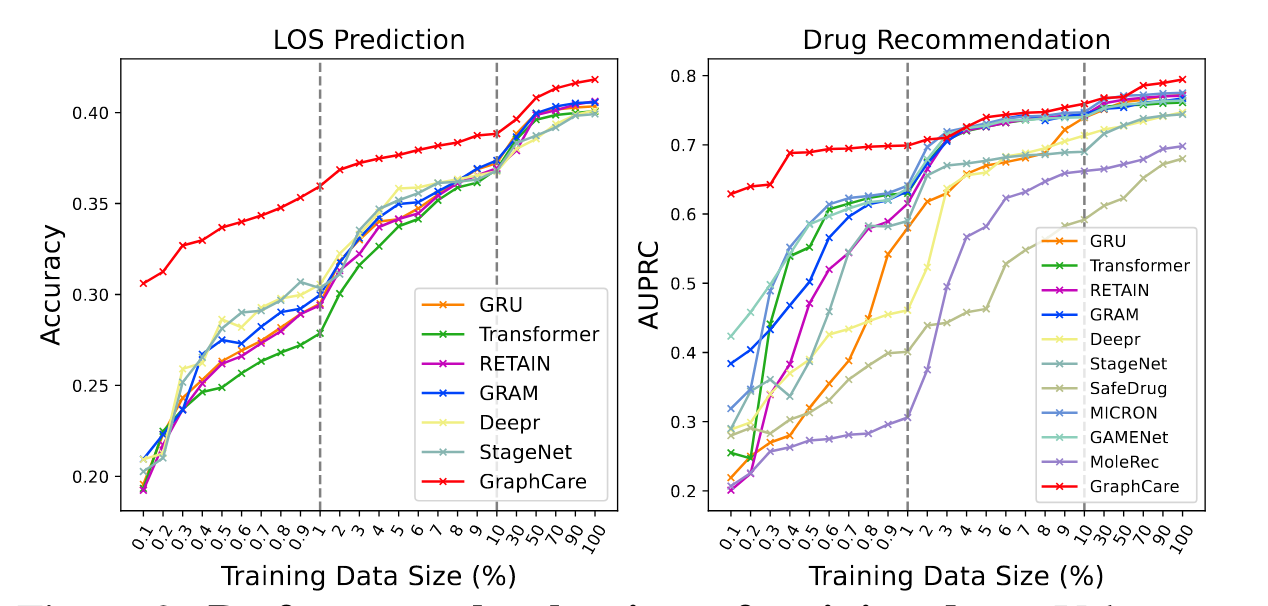
图8. 训练数据量影响
KG规模的影响
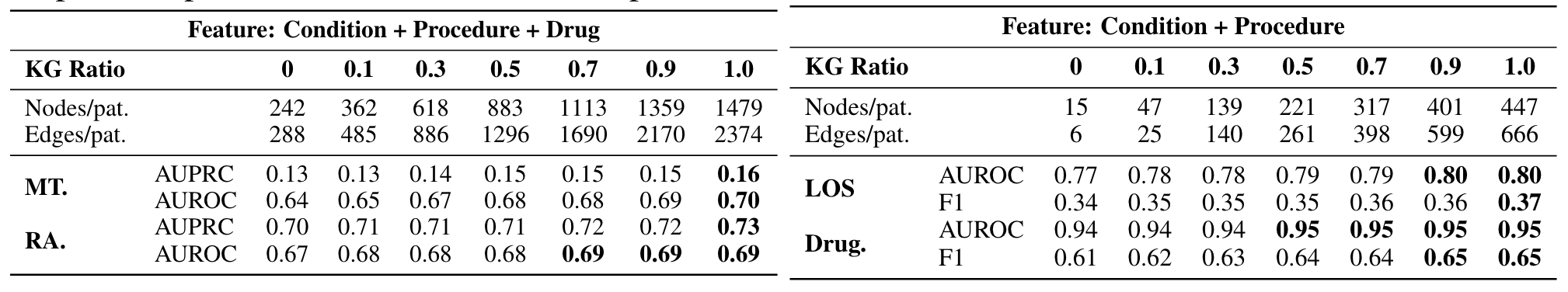
图9. KG规模影响
患者的不同表示效果
- 绿色:zigraph; 蓝色:zinode; 红色:zijoint
- 不同任务有差别,但整体上拼接2个表示会更好
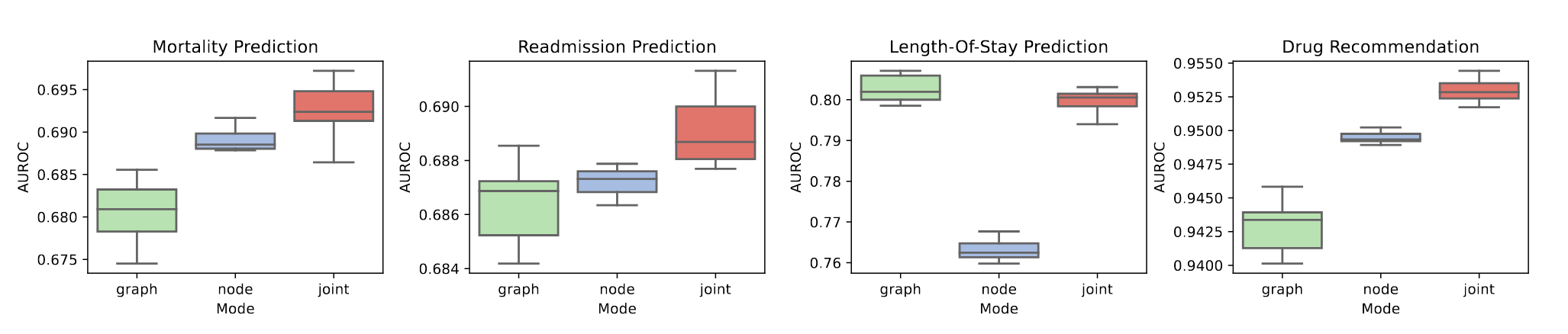
图10. 使用不同患者表示的结果差别
解释性分析
图11 给了被正确预测为死亡的患者,a 图中重要的节点有 “deadly cancer”,“life-threatening"等。b 图显示了患者直接相关的实体,如"bronchiectasis”(支气管扩张)和"pneumonia"(肺炎)。c, d, e 图则展开这些重要节点的相关节点信息。
用Gephi画的图:https://gephi.org/
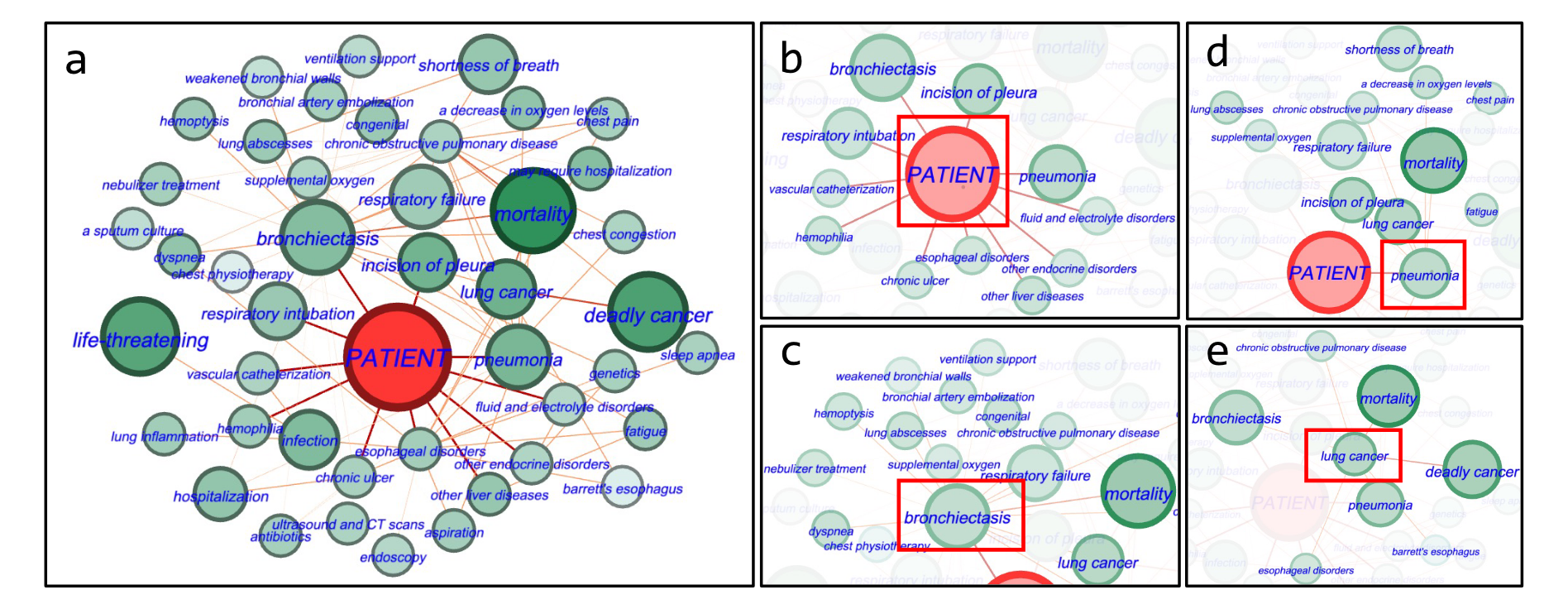
图11. 解释性分析
更多细节和结果
见文章附录,包括:
- 数据预处理的一些细节
- 将患者当作个性化KG的一个节点还是当作一个graph?
- 超参数选择
代码
稍有点复杂,需结合PyHealth库3一起看
总结
本文提出了利用个性化知识图谱进行健康预测的方法GraphCare, 主要有以下几点贡献/创新:
- 从LLM中提取医疗知识
- 将患者的就诊记录转换成个性化的知识图谱
- 设计了双attention机制的BAT
我的想法:现在是按照方法的逻辑一步步往前推,强调做了什么。也许改成出于某个考虑或者为了什么目标,所以选择了哪个策略更方便理解。
链接🔗
1 https://www.nlm.nih.gov/research/umls/index.html
2 https://mimic.mit.edu/docs/iii/tables/
3 https://pyhealth.readthedocs.io/en/latest/